南�v所��Z��环境DNA技术解析河口鱼�cȝ��落时�I�差异研�I�取得新�q�展
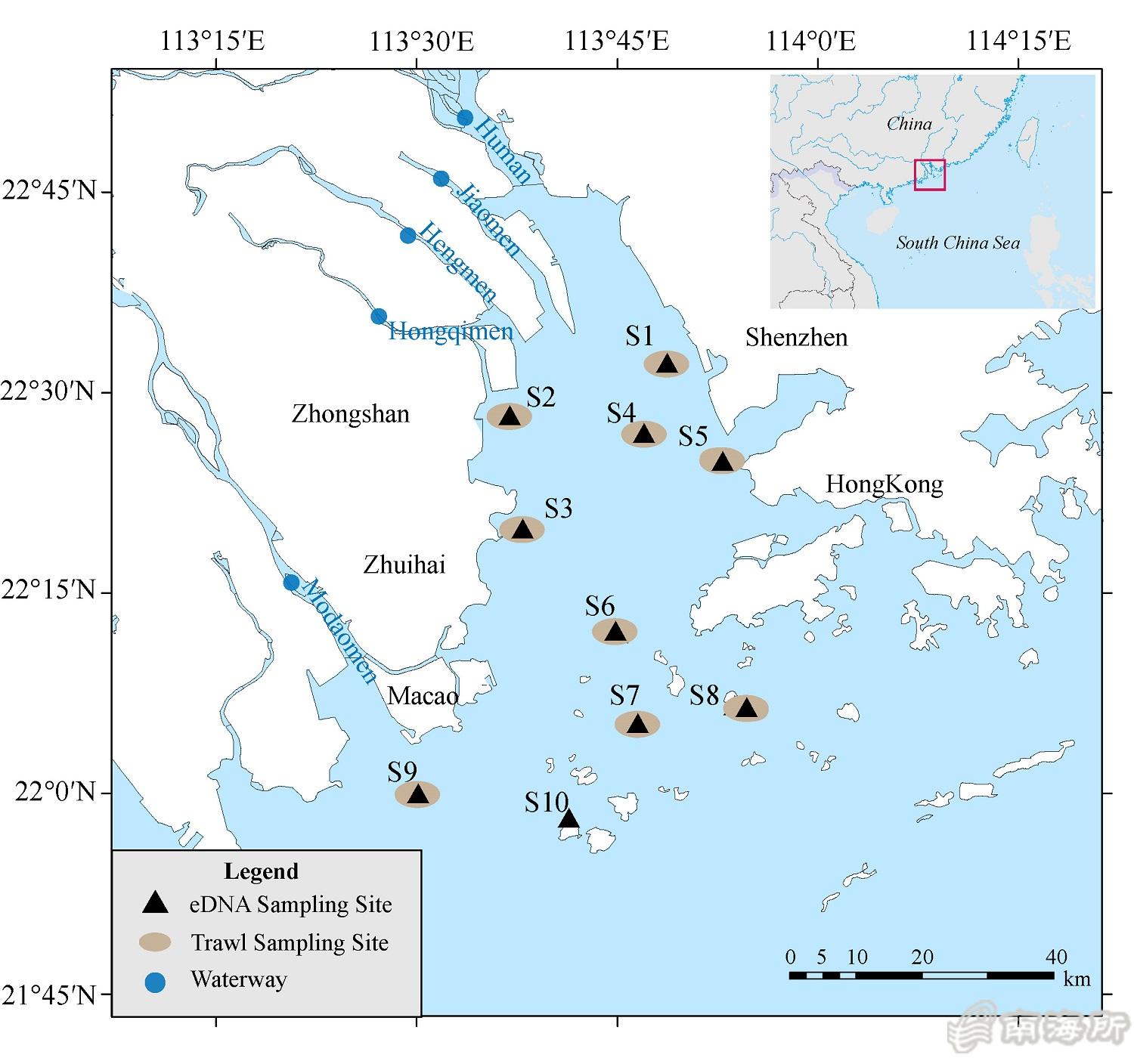
研究区域珠江口采样位点示意图
�q�日�Q�中国水产科学研�I����南�v水��研究所南�v渔业资源调查与评估团队和南�v珍稀�Ȓ危动物保护团队在利用环境DNA�Q�eDNA�Q�技术解析河口鱼�c�d����h��和鱼类���落时空差异斚w��取得新进展。该研究成果论文以�?span style="font-family: "times new roman";">Comparison of environmental DNA metabarcoding and bottom trawling for detecting seasonal fish communities and habitat preference in a highly disturbed estuary”�ؓ题发表在国际生态学领域期刊Ecological Indicators�Q�JCR Q1�Q�上。蒋佩文为第一作者,陈作志与李敏为通讯作者�?/p>
沛_��鱼类是河口生态系�l�的重要�l�成部分�Q�在人类�z�d��日益加剧的背景下�Q�鱼�c�d����h��和���落�l�构的监���越来越凸显光���要性。然而河口�v域水��p�����、航�U�较多和水体���浊�{�,现有的一些调查手�D�常常受��C��些限制。研�I�团队在之前建立的河口�v域eDNA富集�Ҏ���Q�DOI: 10.24272/j.issn.2095-8137.2021.331�Q�DOI: 10.12131/20210304�Q�和珠江沛_��本底鱼类eDNA宏条形码数据库(DOI: 10.12131/20210210�Q�的基础上,利用eDNA宏条形码研究了珠江口伶仃�z�的鱼类物种�l�成、群落时�I�差异以及与环境因子的关�p�,�q�与底拖�|�调查方法进行了比较�?/p>
该研�I�于�U�季和春季在珠江沛_���?0个监���点同时�q�行了底拖网捕捞、水体采样和环境因子���量。水体样本过滤后提取eDNA�Q�利用MiFish-U/E引物�q�行�U�粒�?2SrRNA基因�Q�约172bp�Q�的扩增、徏库、测序和OTU注释�Q�通过数据库的比对获得鱼类物种�l�成。主要结果有�Q?. eDNA�Ҏ��在每个采��L��均比底拖�|�明显检出更多的物种�Q���M��上eDNA共检�?14�U�鱼�c�(底拖�|?0�U�)�Q�同时eDNA������种�cȝ��相对丰度与鱼�cȝ���U�丰�?生物量之间存在显著相��x��。鱼�cȝ��态习性分析表明,eDNA可以���出因传统渔网�|�目大小限制而漏���的小型鱼�c�,佉K���cȝ��落生态特征更加完��_��2. 两种�Ҏ��均检���到鱼类�U�类的季节更替,��x��季和�U�季鱼类���落存在显著差异�Q�eDNA�Ҏ��������到的显著性比底拖�|�更强�?. eDNA与底拖网一样均揭示了鱼�cȝ��落组成差异随着地理距离的增加而显著增加。上�q�结果表明在沛_��生态系�l�中�Q�特别是传统调查�Ҏ��存在困难的情况下�Q�eDNA宏条形码可作��Z���U�重要的补充手段甚至替代�Ҏ��应用于鱼�cȝ��落的季节性变化和�I�间分布变化的监���,提升评估沛_��生态系�l�中鱼类���落�l�构的能力,在渔业研�I����域和生态环境监���领域具有很好的应用前景�?/p>
该研�I�得到国安���点研发计划(2018YFD0900906�Q�、中国水产科学研�I����中央�U�公益性科研院所基本�U�研业务费(2020TD05�?021SD01�?021SD18�Q�等��目资助�?/p>
论文链接�Q?a >https://www.sciencedirect.com/science/article/pii/S1470160X22012274
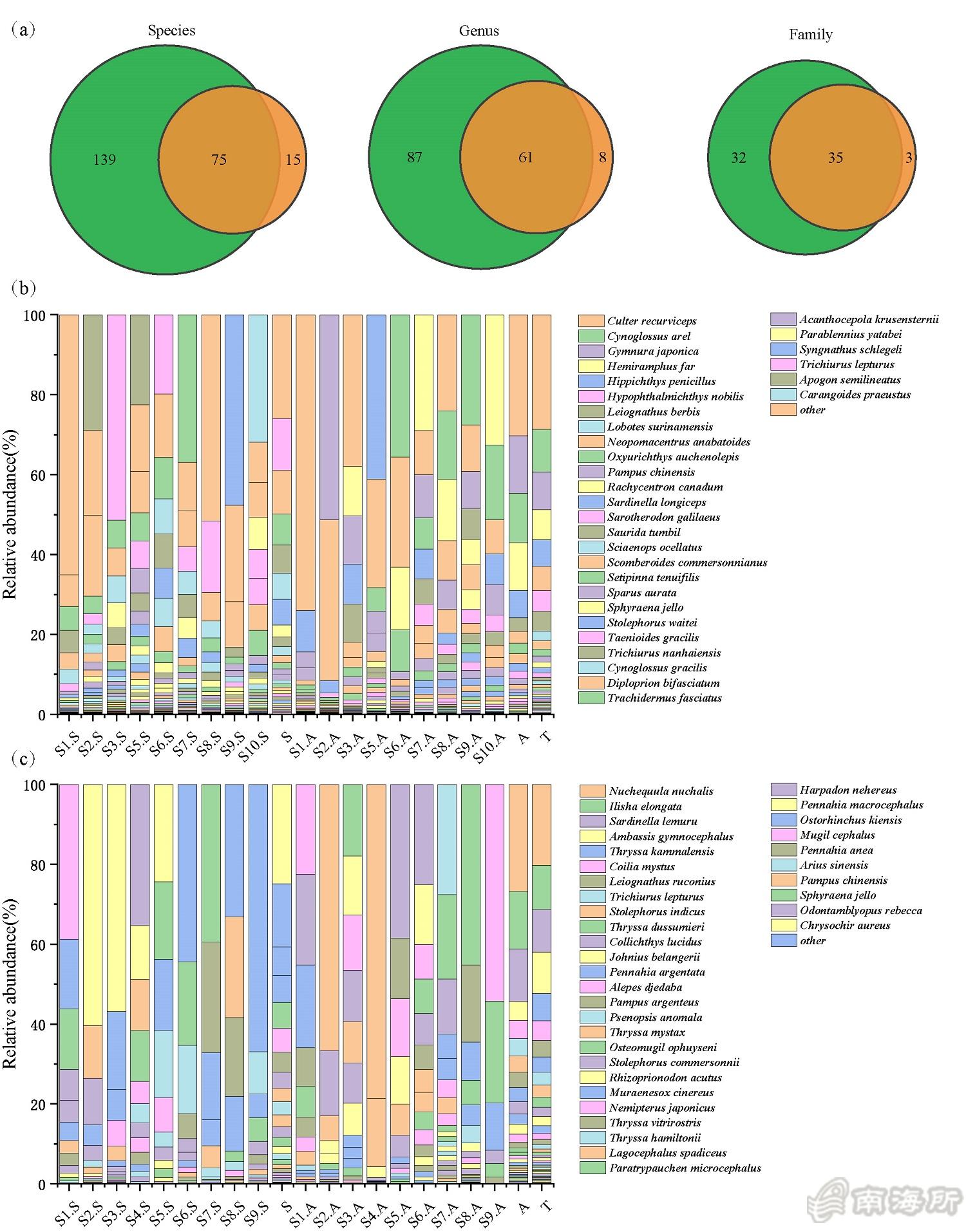
��Z��eDNA和底拖网的鱼�cȝ���U�组成对�?/p>
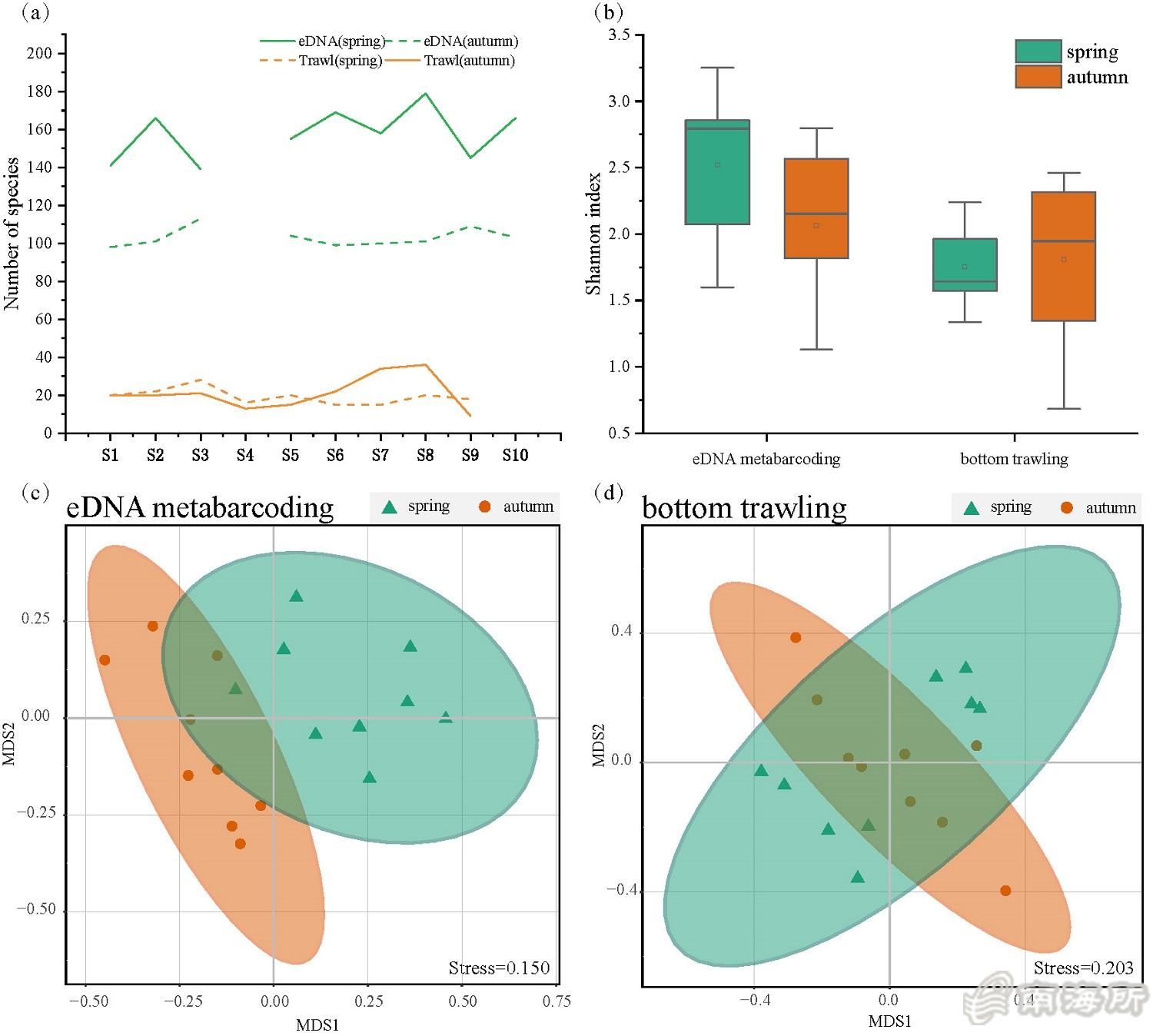
eDNA和底拖网������的物种数量、α多��h��和NMDS 排序



 �_�公�|�安�?4010502001742�?/a>
�_�公�|�安�?4010502001742�?/a>